class: middle, inverse, title-slide .title[ # Single-cell RNA sequencing ~ Session 2 ] .subtitle[ ## <html><br /> <br /> <hr color='#EB811B' size=1px width=796px><br /> </html><br /> Bioinformatics Resource Center - Rockefeller University ] .author[ ### <a href="https://rockefelleruniversity.github.io/SingleCell_Bootcamp/" class="uri">https://rockefelleruniversity.github.io/SingleCell_Bootcamp/</a> ] .author[ ### <a href="mailto:brc@rockefeller.edu" class="email">brc@rockefeller.edu</a> ] --- ## Set Up All prerequisites, links to material and slides for this course can be found on github. At this point hopefully everyone has installed the required packages listed on our homepage. * [Bootcamp](https://rockefelleruniversity.github.io/SingleCell_Bootcamp/) Or can be downloaded as a zip archive from here. * [Download zip](https://github.com/rockefelleruniversity/SingleCell_Bootcamp/zipball/master) * Many of the raw and intermediate files can be found on [DropBox](https://www.dropbox.com/scl/fo/1k4y3fvfbissu1vds5kru/AN-CDAVfqa7H5pJT4wCchj0?rlkey=xi9fvilmqjt5j5er73cbocnui&st=zwlvcf6e&dl=0). --- ## Course materials Once the zip file in unarchived. All presentations as HTML slides and pages, their R code and HTML practical sheets will be available in the directories underneath. * **presentations/slides/** Presentations as an HTML slide show. * **presentations/singlepage/** Presentations as an HTML single page. * **presentations/r_code/** R code in presentations. * **exercises/** Practicals as HTML pages. * **answers/** Practicals with answers as HTML pages and R code solutions. --- ## A basic scRNAseq workflow  --- ## A basic scRNAseq workflow - Load data into Seurat - Data normalization with Seurat - SCT normalization - Dimension reduction and clustering with Seurat - CellBender and other tools for ambient RNA correction - Calculate mitochondrial content - Estimate cell cycle phases with Seurat and cyclone - Evaluate Doublets with scrublet - QC plots - Filtering, regression and review - Evaluate results - cell type specific clusters - Identify marker genes by clusters - Loupe Browser --- ## Seurat * An R toolkit for single cell genomics [link](https://satijalab.org/seurat/index.html) * The workflow we used in this section was orginally based on Seurat vignette [link](https://satijalab.org/seurat/articles/pbmc3k_tutorial.html). * References + **Seurat V5**, Hao et al., *Nature Biotechnology* (2023) [link](https://www.nature.com/articles/s41587-023-01767-y) + **Seurat V4**, Hao, Hao et al., *Cell* (2021) [link](https://doi.org/10.1016/j.cell.2021.04.048) + **Seurat V3**, Stuart, Butler et al., *Cell* (2019) [link](https://www.cell.com/cell/fulltext/S0092-8674(19)30559-8) + **Seurat V2**, Butler, et al., *Nat Biotechnol* (2018) [link](https://doi.org/10.1038/nbt.4096) + **Seurat V1**, Satija, Farrell, et al., *Nat Biotechnol* (2015) [link](https://doi.org/10.1038/nbt.3192) --- ## What is our data? We will use the 8k Human PBMC data from [10X genomics](https://www.10xgenomics.com/datasets/8-k-pbm-cs-from-a-healthy-donor-2-standard-2-1-0). We will be using the filtered dataset that has been produced by Cell Ranger. ``` r download.file("https://cf.10xgenomics.com/samples/cell-exp/2.1.0/pbmc8k/pbmc8k_filtered_gene_bc_matrices.tar.gz", "pbmc8k_filtered_gene_bc_matrices.tar.gz") ``` ``` r untar("pbmc8k_filtered_gene_bc_matrices.tar.gz") ``` We can check the contents of the directory with the `dir()` command. We should see our our unpacked directory `filtered_feature_bc_matrix` and the original tar.gz file. ``` r dir() ``` --- class: inverse, center, middle # Cell Ranger to Seurat <html><div style='float:left'></div><hr color='#EB811B' size=1px width=720px></html> --- ## Load cellranger matrix into matrix ``` r mtx_dir <- "filtered_gene_bc_matrices/GRCh38" ``` ``` r library(Seurat) mtx <- Seurat::Read10X(mtx_dir) ``` ``` r is(mtx) ``` ``` ## [1] "dgCMatrix" "CsparseMatrix" "dsparseMatrix" "generalMatrix" ## [5] "AnyMatrix" "V3Matrix" "dMatrix" "sparseMatrix" ## [9] "compMatrix" "Matrix" "replValueSp" ``` ``` r head(mtx) ``` ``` ## 6 x 8381 sparse Matrix of class "dgCMatrix" ``` ``` ## [[ suppressing 8381 column names 'AAACCTGAGCATCATC-1', 'AAACCTGAGCTAACTC-1', 'AAACCTGAGCTAGTGG-1' ... ]] ``` ``` ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 1 . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . 1 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . 1 . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . 1 . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . 1 . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . 1 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . 1 . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . 1 . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . 1 . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . 1 . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . 1 . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . 1 . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . 1 . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . 1 . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . 1 . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . 1 . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . 1 . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . 1 . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . 1 . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . 1 . . . . . . . . . . . . . 1 . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . 1 . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . 1 . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . 1 . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . 1 . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## ## RP11-34P13.3 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## FAM138A . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## OR4F5 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.7 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.8 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ## RP11-34P13.14 . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . ``` --- ## Load in .h5 format matrix As an alternative we can use *Read10X_h5* function to read cellranger matrix from an **.h5** file. This format is often used by python packages. ``` r h5_file <- "path to matrix h5 file" h5_file <- "~/Downloads/pbmc8k_raw_gene_bc_matrices_h5.h5" mtx <- Seurat::Read10X_h5(h5_file) ``` --- ## Create Seurat object from matrix The matrix is loaded into Seurat object with **CreateSeurtObject()** Cut-offs: + min_gene: minimum genes detected per cell: *cells with too few genes detected* + min_cell: minimum cells a gene expressed in: *Remove genes expressed in too few cells* ``` r sample_id <- "PBMC_8k" # sample name min_gene <- 200 min_cell <- 10 ``` ``` r seu_obj <- Seurat::CreateSeuratObject(mtx, project = sample_id, min.cells = min_cell, min.features = min_gene) ``` --- ## Add sample names to Seurat object We typically add sample information as this will add clarity later when we think about multi-sample comparisons. You will notice throughout this workflow we will keep assigning back to the same Seurat object as we update and modify the object as we process our data. ``` r seu_obj[["dset"]] <- sample_id # Create a category for sample seu_obj <- Seurat::RenameCells(seu_obj, add.cell.id = sample_id) # add sample name in front of cell barcode ``` --- ## The Seurat object We can look at the Seurat object to get information about the dataset. ``` r seu_obj ``` ``` ## An object of class Seurat ## 15446 features across 8381 samples within 1 assay ## Active assay: RNA (15446 features, 0 variable features) ## 1 layer present: counts ``` ``` r head(seu_obj, 2) ``` ``` ## orig.ident nCount_RNA nFeature_RNA dset ## PBMC_8k_AAACCTGAGCATCATC-1 PBMC_8k 2391 868 PBMC_8k ## PBMC_8k_AAACCTGAGCTAACTC-1 PBMC_8k 1694 806 PBMC_8k ``` --- class: inverse, center, middle # Normalization, feature selection, and data scaling <html><div style='float:left'></div><hr color='#EB811B' size=1px width=720px></html> --- ## To scaled data * Normalization: Feature counts are normalized to total counts each cell * Feature selection: Identifies features that are highly variable * Data scaling: Scales and centers features in the dataset --- ## Log normalization To do this in Seurat we have 3 functions: * log normalization with *NormalizeData()* * Identify Variable Features with *FindVariableFeatures()* * Scale Data with *ScaleData()* --- ## Log normalization - Normalization When using the *NormalizeData()* function we use the defaults for the normalization method and scale factor. ``` r seu_obj <- NormalizeData(seu_obj, normalization.method = "LogNormalize") ``` ``` ## Normalizing layer: counts ``` --- ## Log normalization - Features Next we use *FindVariableFeatures()* with the default VST method. We can select the number features we want to identify. These will be the most variable. In this case we are taking 3000. ``` r seu_obj <- FindVariableFeatures(seu_obj, select.method = "vst", nfeatures = 3000) ``` ``` ## Finding variable features for layer counts ``` --- ## Log normalization - Scaling Scales and center features in the dataset ``` r seu_obj <- ScaleData(seu_obj) ``` ``` ## Centering and scaling data matrix ``` --- ## Highly Variable Features - Highly variable features were colored with *red*. - Gene symbols of the top 10 highly variable features were labeled near by the spots ``` r top10 <- head(VariableFeatures(seu_obj), 10) plot1 <- VariableFeaturePlot(seu_obj) plot2 <- LabelPoints(plot = plot1, points = top10, repel = TRUE) plot2 ``` --- ## Highly Variable Features - Highly variable features were colored with *red*. - Gene symbols of the top 10 highly variable features were labeled near by the spots ``` ## When using repel, set xnudge and ynudge to 0 for optimal results ``` <!-- --> --- ## SCTransform  [overview](./imgs/scRNA_workflow_ver001_20231017.png) --- ## SCTransform In our simplified workflow we used a log normalization approach. Recently an alternative approach has been gaining popularity: SCTransform. Lets compare this back to our log normalization. --- ## SCTransform - SCTransform is a normalization based on negative binomial regression. - It is performed with **SCTransform()** function of Seurat. - This function equal to the combinations of NormalizeData(), FindVariableFeatures(), and ScaleData(). ``` r seu_obj <- SCTransform(seu_obj, variable.features.n = 3000) seu_obj ``` ``` ## An object of class Seurat ## 30892 features across 8381 samples within 2 assays ## Active assay: SCT (15446 features, 3000 variable features) ## 3 layers present: counts, data, scale.data ## 1 other assay present: RNA ``` --- ## SCTransform The normalized data is stored in assay **SCT**. We can see this is now the active assay. The log results are stored in the **RNA** assay. We can control which is the active assay with the *DefaultAssay()* function. ``` r DefaultAssay(seu_obj) <- "RNA" DefaultAssay(seu_obj) <- "SCT" ``` --- ## SCTransform vs log normalization We can extract out the results of our normalized and scaled data with the *GetAssayData()* function. ``` r log_mat <- GetAssayData(seu_obj, assay = "RNA", slot = "data") log_mat <- as.matrix(log_mat) log_avgExp <- rowMeans(log_mat) sct_mat <- GetAssayData(seu_obj, assay = "SCT", slot = "data") sct_mat <- as.matrix(sct_mat) sct_avgExp <- rowMeans(sct_mat) ``` --- ## log normalization vs SCTransform We can test the concordance with Spearman's correlation. ``` r library(ggplot2) dat <- data.frame(logNorm = log_avgExp, SCT = sct_avgExp) cor_val <- cor.test(log_avgExp, sct_avgExp, method = "spearman") ggplot(dat, aes(x = logNorm, y = SCT)) + geom_point() + geom_smooth() + labs(x = "Log_Normalization", y = "SCTransform", subtitle = paste0("rho=", round(cor_val$estimate, 3), "; p-value=", cor_val$p.value[1])) + theme_classic() ``` --- ## log normalization vs SCTransform We can see there is very high similarity between the results. ``` ## Warning in cor.test.default(log_avgExp, sct_avgExp, method = "spearman"): ## Cannot compute exact p-value with ties ``` ``` ## `geom_smooth()` using method = 'gam' and formula = 'y ~ s(x, bs = "cs")' ``` <!-- --> --- class: inverse, center, middle # Dimension Reduction <html><div style='float:left'></div><hr color='#EB811B' size=1px width=720px></html> --- ## Dimension Reduction At this point, in theory we should have a nice clean dataset, so we can start pushing towards the biological questions in our dataset. scRNAseq is highly dimensional data. To be able to tackle our data with approaches like clustering we need to simplify it down with dimension reduction. There are several steps: - Perform linear dimensional reduction with Principle Component Analysis (PCA) - Determine dimensionality with Elbow plot - Process non-linear dimensional reduction with UMAP - Need to select major PCs for the following clustering --- ## Linear dimensional reduction Perform PCA with *RunPCA()* function in Seurat. Set Principle components to 50. ``` r set.seed(1001) DefaultAssay(seu_obj) <- "RNA" seu_obj <- RunPCA(seu_obj, assay = "RNA", npcs = 50) ``` ``` ## PC_ 1 ## Positive: CST3, LYZ, MNDA, CSTA, TYROBP, LST1, FCN1, FTL, AIF1, CTSS ## FCER1G, TYMP, S100A9, RP11-1143G9.4, LGALS1, FTH1, S100A8, LGALS2, SERPINA1, SPI1 ## S100A11, FGL2, PSAP, GRN, CFD, GPX1, AP1S2, MS4A6A, CLEC7A, VCAN ## Negative: LTB, TRAC, RPS18, CD3D, EEF1A1, IL32, TRBC2, CD69, LDHB, CD3G ## IL7R, CD27, CD7, IFITM1, RPS2, CD2, LEF1, CCR7, TRBC1, SPOCK2 ## NOSIP, FLT3LG, LINC00861, CD247, TRAT1, LDLRAP1, CD6, DNAJB1, MAL, CTSW ## PC_ 2 ## Positive: IL32, CD3D, TMSB4X, CTSW, CD7, TRAC, NKG7, GZMA, S100A4, CST7 ## IFITM1, CCL5, GZMM, CD247, ANXA1, PRF1, CD2, KLRD1, CD3G, PFN1 ## SRGN, KLRB1, ITGB2, S100A6, ID2, HOPX, IL7R, LYAR, KLRG1, GNLY ## Negative: CD79A, MS4A1, IGHM, IGHD, CD79B, TCL1A, IGKC, LINC00926, BANK1, CD22 ## CD74, HLA-DQB1, HLA-DRA, HLA-DPA1, VPREB3, TNFRSF13C, FCER2, HLA-DPB1, MEF2C, FAM129C ## RALGPS2, HLA-DRB1, HLA-DOB, HLA-DQA1, FCRLA, EAF2, SPIB, HVCN1, HLA-DMB, TSPAN13 ## PC_ 3 ## Positive: GZMB, NKG7, PRF1, CST7, KLRD1, GZMA, CLIC3, FGFBP2, KLRF1, GNLY ## HOPX, SPON2, C12orf75, CD160, CCL5, RHOC, CTSW, CCL4, GZMH, FCGR3A ## XCL2, MATK, CMC1, TRDC, S1PR5, ADGRG1, APOBEC3G, PLEK, IL2RB, CLIC1 ## Negative: RPLP1, LEF1, CCR7, RPS18, RPS24, RPS2, MAL, S100A12, LDHB, ACTN1 ## EEF1A1, S100A8, VCAN, TRABD2A, JUNB, S100A9, RP11-1143G9.4, CD14, TRAC, LDLRAP1 ## SERINC5, NOSIP, LRRN3, RGCC, LTB, FCN1, IL7R, FOS, RBP7, CSTA ## PC_ 4 ## Positive: LILRA4, SERPINF1, LRRC26, IL3RA, CLEC4C, SCT, PLD4, PPP1R14B, TPM2, DERL3 ## ITM2C, PTCRA, DNASE1L3, LINC00996, TNFRSF21, GAS6, FCER1A, LAMP5, SMPD3, UGCG ## MAP1A, PTPRS, RP11-73G16.2, JCHAIN, MYBL2, CCDC50, SCAMP5, ASIP, IRF7, LILRB4 ## Negative: KLRD1, NKG7, CST7, FGFBP2, PRF1, KLRF1, GZMA, GNLY, CCL4, HOPX ## CD160, MS4A1, IGHD, CD79B, CD79A, SPON2, LINC00926, MT-CO3, XCL2, MT-CO1 ## TRDC, S1PR5, GZMH, FCER2, MATK, ADGRG1, TTC38, FCGR3A, CCL5, CMC1 ## PC_ 5 ## Positive: RPS2, EEF1A1, CDKN1C, HES4, TMSB10, RPS18, SIGLEC10, CSF1R, BATF3, TCF7L2 ## FCGR3A, CORO1A, SLC25A6, LRRC25, RPLP1, MS4A4A, ABI3, YBX1, HCK, WARS ## MS4A7, RNASET2, JUNB, HLA-DQA1, NEURL1, ENHO, CSTB, RP11-1008C21.1, ANXA2, CLIC2 ## Negative: SDPR, PPBP, GNG11, PF4, TUBB1, SPARC, CLU, HIST1H2AC, GP9, ACRBP ## CTTN, TMEM40, C2orf88, CMTM5, ITGA2B, TSC22D1, MAP3K7CL, NRGN, HRAT92, MMD ## CLDN5, PTCRA, HGD, TREML1, HIST1H2BJ, CD9, MYLK, SH3BGRL2, GMPR, LCN2 ``` --- ## Evaluate dimensionality with elbow plot Typically we want to balance having enough/not too many groups with the most variance explained by the groups, to get the most parsimonious results. We use elbow plots to find an inflection point. ``` r ElbowPlot(seu_obj, ndims = 50, reduction = "pca") ``` <!-- --> --- ## Deriving the elbow We can use cutoffs to derive this computationally based on: * How low variation is. * How much variation has already been described. * The rate of change in variation between PCs. ``` r library(dplyr) # Determine percent of variation associated with each PC pct <- seu_obj[["pca"]]@stdev/sum(seu_obj[["pca"]]@stdev) * 100 # Calculate cumulative percents for each PC cumu <- cumsum(pct) ``` --- ## Deriving the elbow We can use cutoffs to derive this computationally based on: * How low variation is. * How much variation has already been described. * The rate of change in variation between PCs. ``` r # Determine which PC exhibits cumulative percent greater than 90% and % # variation associated with the PC as less than 5 co1 <- which(cumu > 90 & pct < 5)[1] # Determine the difference between variation of PC and subsequent PC, last # point where change of % of variation is more than 0.1%. co2 <- sort(which((pct[1:length(pct) - 1] - pct[2:length(pct)]) > 0.1), decreasing = T)[1] + 1 pc <- min(co1, co2) pc ``` ``` ## [1] 17 ``` --- ## Deriving the elbow We can now look at our cutoff on our elbow plot. ``` r plot_df <- data.frame(pct = pct, cumu = cumu, rank = 1:length(pct)) ggplot(plot_df, aes(cumu, pct, label = rank, color = rank > pc)) + geom_text() + geom_vline(xintercept = 90, color = "grey") + geom_hline(yintercept = min(pct[pct > 5]), color = "grey") + theme_bw() ``` --- ## Deriving the elbow We can now look at our cutoff on our elbow plot. This seems to match the point where our data plateaus. <!-- --> --- ## Make non-linear dimensional reduction * Estimate neighbors with **FindNeighors()** function in *Seurat* + reduction method: *PCA* + use dimensions from 1 to the PCs detected by elbow plot * Make non-linear dimensional reduction with UMAP ``` r seu_obj <- FindNeighbors(seu_obj, dims = 1:pc, reduction = "pca") ``` ``` ## Computing nearest neighbor graph ``` ``` ## Computing SNN ``` ``` r seu_obj <- RunUMAP(seu_obj, dims = 1:pc, reduction = "pca") ``` ``` ## Warning: The default method for RunUMAP has changed from calling Python UMAP via reticulate to the R-native UWOT using the cosine metric ## To use Python UMAP via reticulate, set umap.method to 'umap-learn' and metric to 'correlation' ## This message will be shown once per session ``` ``` ## 17:58:05 UMAP embedding parameters a = 0.9922 b = 1.112 ``` ``` ## 17:58:05 Read 8381 rows and found 17 numeric columns ``` ``` ## 17:58:05 Using Annoy for neighbor search, n_neighbors = 30 ``` ``` ## 17:58:05 Building Annoy index with metric = cosine, n_trees = 50 ``` ``` ## 0% 10 20 30 40 50 60 70 80 90 100% ``` ``` ## [----|----|----|----|----|----|----|----|----|----| ``` ``` ## **************************************************| ## 17:58:06 Writing NN index file to temp file /tmp/Rtmpu029jb/file2c824e77d15c ## 17:58:06 Searching Annoy index using 1 thread, search_k = 3000 ## 17:58:08 Annoy recall = 100% ## 17:58:09 Commencing smooth kNN distance calibration using 1 thread with target n_neighbors = 30 ## 17:58:09 Initializing from normalized Laplacian + noise (using RSpectra) ## 17:58:10 Commencing optimization for 500 epochs, with 353242 positive edges ## 17:58:10 Using rng type: pcg ## 17:58:17 Optimization finished ``` --- ## UMAP We can now draw the results of our UMAP, which clearly separates our cells intro different groups. But this doesn't mean anything until we cluster. ``` r DimPlot(seu_obj) ``` <!-- --> --- ## Why UMAPs? We will be making a lot of UMAPs going forward. Relatively new approach (2018) and is generally used more than tSNE now. Though it sometimes faces criticism we find it does a good job on most data sets. It allows a comprehensible 2D visualization of your result. For a detailed explanation and discussion of dimension reduction approaches here are some resources: [Data Science blog post](https://towardsdatascience.com/dimensionality-reduction-for-data-visualization-pca-vs-tsne-vs-umap-be4aa7b1cb29) [Bioinformatic presentation - Babraham Institute](https://www.bioinformatics.babraham.ac.uk/training/10XRNASeq/Dimension%20Reduction.pdf) --- ## A basic scRNAseq workflow  --- ## Clustering - The cells are then clustered using *FindClusters()* function in Seurat. - The clustering is differentiated by **original Louvain algorithm**. - To see different options for clustering with Seurat, you can alter the parameter: algorithm. If you want to learn about which algorithms are available, you can quickly read the Seurat manual by typing ?Seurat in R --- ## Identify clusters We will have a quick look first at how to run the clustering, using the *FindClusters()* function. We just need to provide our Seurat object and a resolution. ``` r seu_obj <- FindClusters(seu_obj, resolution = 0.5) ``` ``` ## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck ## ## Number of nodes: 8381 ## Number of edges: 314488 ## ## Running Louvain algorithm... ## Maximum modularity in 10 random starts: 0.9170 ## Number of communities: 15 ## Elapsed time: 0 seconds ``` ``` r seu_obj[["cluster_byDefault"]] <- seu_obj$seurat_clusters ``` --- ## Identify clusters - Now you can view your default clusters with the DimPlot() function. ``` r DimPlot(seu_obj, group.by = "cluster_byDefault") ``` <!-- --> --- ## A basic scRNAseq workflow  --- class: inverse, center, middle # Droplet processing <html><div style='float:left'></div><hr color='#EB811B' size=1px width=720px></html> --- ## Droplet processing  [overview](./imgs/scRNA_workflow_ver001_20231017.png) --- ## Droplet processing Often empty droplets can still make their way past Cell Ranger, or we might see an issue in the Cell Ranger web report itself (or in downstream steps). If this happens we might want to do custom filtering outside of Cell Ranger. One cause of these issues could be ambient RNAs. Ambient RNAs from lysed cells make empty droplets seem like they contain a cell or contaminate droplets that already have a cell. Lets look at the PBMC 8k data from 10X Genomics as an example to deal with these issues. This time we will use the raw matrix. This way we don't use the filtering that Cell Ranger does. --- ## CellBender CellBender is a command line toolkit developed to eliminate technical artifacts from scRNA-Seq data such as empty droplets and ambient RNAs [link](https://cellbender.readthedocs.io/en/latest/introduction/index.html). In the current version, it contains remove-background module, which is applied to: * Detect empty droplets and ambient RNAs from raw count matrix in CellRanger output. * Remove any droplets considered to be empty, correct the interference of ambient RNAs. * CellBender is based on machine-learning strategy. It's quite time-consuming. We can speed up the processing by using CUDA version of CellBender. However, this means you will need access to a GPU. --- ## CellBender command We take the raw matrices from Cell Ranger and run CellBender. The cuda argument will speed up the function, but requires your run it on a GPU. [This would be run through Terminal on a HPC] ``` sh input_h5=the_raw_matrix_in_h5_format_from_cellranger #essential output_h5=assign_the_h5_file_path_for_the_cellbender_corrected_matrix # essential fpr=threshold_of_FALSE_POSITIVE_RATE # default 0.01 epochs=number_of_epochs_to_train # default 150 num_train=number_of_times_to_attempt_to_train_the_model # default 1. would speed up while setting greater cellbender remove-background --input $input_h5 --output $output_h5 --fpr $fpr --epochs $epochs --num-training-tries $num_train --cuda ``` --- ## CellBender and Seurat The h5 file produced by CellBender is currently incompatible with Seurat. We can use the following function to convert the h5 file to a Seurat object. CellBender has a guide how to convert the h5 file to a Seurat object [here](https://cellbender.readthedocs.io/en/latest/tutorial/index.html#open-in-seurat), using PyTables. This is another command line tool. ``` sh ptrepack --complevel 5 celbender_filtered.h5:/matrix celbender_filtered_forseurat.h5:/matrix ``` --- ## Cell Ranger vs. CellBender We have some processed results here from CellBender. We can compare this to our filtered matrix from Cell Ranger. ``` r cbFilt_mtx <- Read10X_h5("~/Downloads/cbFilt_PBMC8K_20250131_filtered_forseurat.h5") ``` --- ## Create some functions We are going to try out a few tools and approaches. We want to wrap some of our analysis steps into a function to simplify rerunning things. Normalization: * Log normalization with scale factor = 10,000 (this is approx. the avg # of counts in a cell for 10X scRNA-seq data) * Find Variable features with vst, select top 2000 variable features ``` r data_proc <- function(seu) { seu <- NormalizeData(seu, normalization.method = "LogNormalize", scale.factor = 10000) seu <- FindVariableFeatures(seu, select.method = "vst", nfeatures = 2000) return(seu) } ``` --- ## Create some functions Make clusters: * Scale data with *ScaleData()* * Principle Component Analysis by using *RunPCA()* with npcs=30 PCs * Make non-linear dimensional reduction in UMAP by using *RunUMAP()* with dims=1:10 * Estimate Neighbors by using *FindNeighbors()* with dims=1:10 * Identify clusters with *FindClusters()* by using resolution=0.5 ``` r quick_clust <- function(seu) { set.seed(42) seu <- RunPCA(seu, npcs = 30, verbose = FALSE) seu <- RunUMAP(seu, reduction = "pca", dims = 1:10, verbose = FALSE) seu <- FindNeighbors(seu, reduction = "pca", dims = 1:10, verbose = FALSE) seu <- FindClusters(seu, resolution = 0.5, verbose = FALSE) return(seu) } ``` --- ## Data processing ``` r message("processing matrix from CellBender") seu <- CreateSeuratObject(cbFilt_mtx) seu <- data_proc(seu) seu <- ScaleData(seu) seu <- quick_clust(seu) seu_cbFilt <- seu ``` --- ## Evaluate with marker gene expression - Without CellBender ``` r DimPlot(seu_obj, group.by = "seurat_clusters", pt.size = 0.1, label = TRUE) + NoLegend() ``` <!-- --> --- ## Evaluate with marker gene expression - With CellBender ``` r DimPlot(seu_cbFilt, group.by = "seurat_clusters", pt.size = 0.1, label = TRUE) + NoLegend() ``` <!-- --> --- ## Evaluate with marker gene expression - We will check the expression of some monocyte markers: LYZ and HLA-DRA (present in B-cells also) - Without CellBender ``` r mark_gene <- c("LYZ", "HLA-DRA") FeaturePlot(seu_obj, features = mark_gene, pt.size = 0) ``` <!-- --> --- ## Evaluate with marker gene expression - Without CellBender ``` r VlnPlot(seu_obj, features = mark_gene, group.by = "seurat_clusters", pt.size = 0) ``` <!-- --> --- ## Evaluate with marker gene expression - With CellBender ``` r FeaturePlot(seu_cbFilt, features = mark_gene, pt.size = 0) ``` <!-- --> --- ## Evaluate with marker gene expression - With CellBender ``` r VlnPlot(seu_cbFilt, features = mark_gene, group.by = "seurat_clusters", pt.size = 0) ``` <!-- --> ``` ## used (Mb) gc trigger (Mb) max used (Mb) ## Ncells 5342027 285.3 9664890 516.2 9664890 516.2 ## Vcells 65129444 496.9 261680288 1996.5 278961202 2128.4 ``` --- ## Other ambient RNA removal methods - There are other popular software used for ambient RNA removal, most which you can use in R - We have code for running soupX in a previous version of this [course](https://github.com/RockefellerUniversity/SingleCell_Bootcamp/releases/tag/v1.0). - Here are the pros and cons of each method:  --- ## Using CellBender object - Let's re-add sample information to the new CellBender object we'll be using from now on ``` r sample_id <- "PBMC_8k" seu_cbFilt[["dset"]] <- sample_id # Create a category for sample seu_cbFilt <- Seurat::RenameCells(seu_cbFilt, add.cell.id = sample_id) # add sample name in front of cell barcode ``` --- ## Droplet processing  [overview](./imgs/scRNA_workflow_ver001_20231017.png) --- class: inverse, center, middle # Mitochondrial Proportion <html><div style='float:left'></div><hr color='#EB811B' size=1px width=720px></html> --- ## Mitochondrial Proportion Like everything scRNAseq is imperfect. Often cellular debris makes its way into a droplet. We can find a lot of these bad droplets by looking for a high percentage of mitochondrial counts. --- ## Estimate mitochondrial proportion The *PercentageFeatureSet()* function can be used to estimate the percentage of all counts that belong to a set of features. These features can be given as a vector or as a pattern. If we give a vector we have to make sure the given genes are detected in the Seurat object. They may have been filtered out. ``` r mt_gene <- c("MT-ND1", "MT-ND2", "MT-ND3", "MT-ND4", "MT-ND4L", "MT-ND5", "MT-ND6", "MT-CO1", "MT-CO2", "MT-CO3", "MT-ATP6", "MT-ATP8", "MT-CYB") mt_gene_det <- mt_gene[mt_gene %in% rownames(seu_cbFilt)] seu_cbFilt[["percent.mt"]] <- PercentageFeatureSet(seu_cbFilt, features = mt_gene_det) summary(seu_cbFilt$percent.mt) ``` ``` ## Min. 1st Qu. Median Mean 3rd Qu. Max. ## 0.000 2.148 2.765 3.985 3.764 100.000 ``` --- ## Estimate mitochondrial proportion We can also use regular expression pattern matching to define our feature sets. This is often easier, but may not be possible, depending on your organism. ``` r seu_cbFilt[["percent.mt2"]] <- PercentageFeatureSet(seu_cbFilt, pattern = "^MT-") summary(seu_cbFilt$percent.mt2) ``` ``` ## Min. 1st Qu. Median Mean 3rd Qu. Max. ## 0.000 2.148 2.765 3.985 3.764 100.000 ``` --- ## Estimate mitochondrial proportion We can double check that this matches easily by comparing the percentages. ``` r library(ggplot2) dat <- data.frame(byPattern = seu_cbFilt$percent.mt, byGene = seu_cbFilt$percent.mt2, stringsAsFactors = FALSE) cor_val <- cor.test(dat$byPattern, dat$byGene, method = "spearman") ggplot(dat, aes(x = byPattern, y = byGene)) + geom_point() + geom_smooth() + labs(x = "% of MT, est by genes", y = "% of MT,est by pattern", subtitle = paste0("rho=", round(cor_val$estimate, 3), "; p-value=", cor_val$p.value[1])) + theme_classic() ``` --- ## Estimate mitochondrial proportion ``` ## Warning in cor.test.default(dat$byPattern, dat$byGene, method = "spearman"): ## Cannot compute exact p-value with ties ``` ``` ## `geom_smooth()` using method = 'gam' and formula = 'y ~ s(x, bs = "cs")' ``` <!-- --> --- class: inverse, center, middle # Cell Cycle Phases <html><div style='float:left'></div><hr color='#EB811B' size=1px width=720px></html> --- ## Cell Cycle Phases The cell cycle can have a massive impact on gene expression variation across a population and is rarely a signature we are trying to find in our data. We can use several methods to regress out these signatures. But, before we do that we need to estimate the cell cycle phase of every cell. --- ## Cell Cycle Phases In this step, we will introduce two methods to estimate cell cycle phase for each cell. - The CellCycleScoring function in *Seurat* - Cyclone function in *scran* --- ## Cell Cycle and Seurat To estimate the cell cycle phase we need a list of genes that correspond to each cell cycle phase. Seurat does provide these in a list called *cc.genes*, which contains S-phase and G2/M-phase genes for humans. ``` r feat_s <- cc.genes$s.genes feat_g2m <- cc.genes$g2m.genes feat_s ``` ``` ## [1] "MCM5" "PCNA" "TYMS" "FEN1" "MCM2" "MCM4" ## [7] "RRM1" "UNG" "GINS2" "MCM6" "CDCA7" "DTL" ## [13] "PRIM1" "UHRF1" "MLF1IP" "HELLS" "RFC2" "RPA2" ## [19] "NASP" "RAD51AP1" "GMNN" "WDR76" "SLBP" "CCNE2" ## [25] "UBR7" "POLD3" "MSH2" "ATAD2" "RAD51" "RRM2" ## [31] "CDC45" "CDC6" "EXO1" "TIPIN" "DSCC1" "BLM" ## [37] "CASP8AP2" "USP1" "CLSPN" "POLA1" "CHAF1B" "BRIP1" ## [43] "E2F8" ``` ``` r feat_g2m ``` ``` ## [1] "HMGB2" "CDK1" "NUSAP1" "UBE2C" "BIRC5" "TPX2" "TOP2A" ## [8] "NDC80" "CKS2" "NUF2" "CKS1B" "MKI67" "TMPO" "CENPF" ## [15] "TACC3" "FAM64A" "SMC4" "CCNB2" "CKAP2L" "CKAP2" "AURKB" ## [22] "BUB1" "KIF11" "ANP32E" "TUBB4B" "GTSE1" "KIF20B" "HJURP" ## [29] "CDCA3" "HN1" "CDC20" "TTK" "CDC25C" "KIF2C" "RANGAP1" ## [36] "NCAPD2" "DLGAP5" "CDCA2" "CDCA8" "ECT2" "KIF23" "HMMR" ## [43] "AURKA" "PSRC1" "ANLN" "LBR" "CKAP5" "CENPE" "CTCF" ## [50] "NEK2" "G2E3" "GAS2L3" "CBX5" "CENPA" ``` --- ## Cell Cycle and Seurat The *CellCycleScoring()* function the scores each cell based on specific features for S-phase/G2M-phase. For a given cell with significant high S.Score or G2M.Score they are assigned as S/G2M. Cells with low both S.Score and G2M.Score were assigned as G1. This will *overestimate* cells in S-/G2M-phases in the tissues with low cell cycle i.e. neurons. We will be focusing on our log normalized results, so first we need to make that the active assay. ``` r DefaultAssay(seu_cbFilt) <- "RNA" seu_cbFilt <- CellCycleScoring(seu_cbFilt, s.features = feat_s, g2m.features = feat_g2m) ``` ``` ## Warning: The `slot` argument of `GetAssayData()` is deprecated as of SeuratObject 5.0.0. ## i Please use the `layer` argument instead. ## i The deprecated feature was likely used in the Seurat package. ## Please report the issue at <https://github.com/satijalab/seurat/issues>. ## This warning is displayed once every 8 hours. ## Call `lifecycle::last_lifecycle_warnings()` to see where this warning was ## generated. ``` ``` ## Warning: The following features are not present in the object: MLF1IP, not ## searching for symbol synonyms ``` --- ## Cell cycle score vs phase To assess our assignment we can check the score given for each cell, against what phase it was assigned to. To do this we need to make a data frame of scores. ``` r dat_s <- data.frame(cell_id = Cells(seu_cbFilt), cat = "S_Score", Phase = seu_cbFilt$Phase, score = seu_cbFilt$S.Score) dat_g2m <- data.frame(cell_id = Cells(seu_cbFilt), cat = "G2M_Score", Phase = seu_cbFilt$Phase, score = seu_cbFilt$G2M.Score) dat <- rbind(dat_s, dat_g2m) dat$Phase <- factor(dat$Phase, levels = c("G1", "S", "G2M")) ``` --- ## Cell cycle score vs phase We can see in both S and G2/M scores are highest in cells with the corresponding assignment. ``` r ggplot(dat, aes(x = Phase, y = score, fill = Phase)) + geom_violin() + labs(x = "", y = "Score", fill = "Phase") + facet_wrap(~cat) + theme_classic() ``` <!-- --> --- ## Cell cycle estimation by cyclone An alternative approach is to use the *cyclone()* function in scran. This is **much slower** than *CellCycleScoring()* in Seurat. The first step is to convert our Seurat object to a SinlgeCellExperiment object. This is a Bioconductor specific format for single cell data. ``` r library(scran) sce <- as.SingleCellExperiment(seu_cbFilt, assay = "RNA") rowData(sce)$SYMBOL <- rownames(sce) ``` ``` r sce ``` ``` ## class: SingleCellExperiment ## dim: 33694 8845 ## metadata(0): ## assays(2): counts logcounts ## rownames(33694): RP11-34P13.3 FAM138A ... AC213203.1 FAM231B ## rowData names(1): SYMBOL ## colnames(8845): PBMC_8k_GCAGTTAGTTCCATGA-1 PBMC_8k_CCTTCCCTCATCTGTT-1 ## ... PBMC_8k_AGCGTATTCGCCAGCA-1 PBMC_8k_GGGTTGCGTCGTCTTC-1 ## colData names(12): orig.ident nCount_RNA ... Phase ident ## reducedDimNames(2): PCA UMAP ## mainExpName: RNA ## altExpNames(0): ``` --- ## Cell cycle estimation by cyclone We also need a list of genes that correspond to each cycle stage. Cyclone does not just assume that unassigned cells are G1. Instead it uses G1-specific genes to actively assign cells to 3 groups, G1, S and G2/M. Cyclone requires a list as an input. The first slot contains the names of each group of genes i.e. G1, S and G2/M. The remaining slots contain the relevant genes for each group. We have the list here for you to load in. ``` r load("data/ccGene_mouse_human_geneSymbol_ensemblID_20220817.RData") ccGene_hs <- ccGene_mm_hs$human_symbol lapply(ccGene_hs, function(x) { head(x, 2) }) ``` ``` ## $G1 ## first second ## 1 GNAI3 PWP1 ## 2 GNAI3 ASF1B ## ## $S ## first second ## 1 GNAI3 SSR4 ## 2 GNAI3 RAB11A ## ## $G2M ## first second ## 1 GNAI3 TSG101 ## 2 GNAI3 CDC6 ``` --- ## Running cyclone We just provide the the SingleCellExperiment object, the cell cycle gene list, and also a vector of all gene names. Remember! This is slow. We have a processed result you can also load in. ``` r assignments <- cyclone(sce, ccGene_hs, gene.names = rowData(sce)$SYMBOL) ``` --- ## Cyclone results Cyclone estimates the score for each phase in each cell. It will then assign the cell cycle phases by the highest score ``` r lapply(assignments, head) ``` ``` ## $phases ## [1] "G1" "G2M" "G1" "G1" "G1" "G1" ## ## $scores ## G1 S G2M ## 1 1.000 0.344 0.000 ## 2 0.006 0.078 0.891 ## 3 1.000 0.097 0.004 ## 4 0.999 0.085 0.012 ## 5 1.000 0.511 0.000 ## 6 0.998 0.056 0.011 ## ## $normalized.scores ## G1 S G2M ## 1 0.744047619 0.25595238 0.000000000 ## 2 0.006153846 0.08000000 0.913846154 ## 3 0.908265213 0.08810173 0.003633061 ## 4 0.911496350 0.07755474 0.010948905 ## 5 0.661813369 0.33818663 0.000000000 ## 6 0.937089202 0.05258216 0.010328638 ``` --- ## Cyclone and Seurat We can take our Cyclone results and assign them back into our orignal Seurat object. ``` r seu_cbFilt[["cyclon_Phase"]] <- assignments$phases seu_cbFilt[["cyclon_G1Score"]] <- assignments$scores$G1 seu_cbFilt[["cyclon_SScore"]] <- assignments$scores$S seu_cbFilt[["cyclon_G2MScore"]] <- assignments$scores$G2M ``` --- ## Cell cycle score vs phase Again, we assess our assignment by checking the score given for each cell, against what phase it was assigned to. We must first make a data frame of scores. ``` r dat_g1 <- data.frame(cell_id = Cells(seu_cbFilt), cat = "cyclon_G1Score", Phase = seu_cbFilt$cyclon_Phase, score = seu_cbFilt$cyclon_G1Score) dat_s <- data.frame(cell_id = Cells(seu_cbFilt), cat = "cyclon_SScore", Phase = seu_cbFilt$cyclon_Phase, score = seu_cbFilt$cyclon_SScore) dat_g2m <- data.frame(cell_id = Cells(seu_cbFilt), cat = "cyclon_G2MScore", Phase = seu_cbFilt$cyclon_Phase, score = seu_cbFilt$cyclon_G2MScore) dat <- rbind(dat_g1, dat_s, dat_g2m) dat$Phase <- factor(dat$Phase, levels = c("G1", "S", "G2M")) dat$cat <- factor(dat$cat, levels = c("cyclon_G1Score", "cyclon_SScore", "cyclon_G2MScore")) ``` --- ## Cell cycle score vs phase We can see in both S and G2/M scores are highest in cells with the corresponding assignment. ``` r ggplot(dat, aes(x = Phase, y = score, fill = Phase)) + geom_violin() + labs(x = "", y = "Score", fill = "Phase") + facet_wrap(~cat) + theme_classic() ``` <!-- --> --- ## Compare the two strategies Cell Cycle Phase determined by *Seurat::CellCycleScoring()* ``` r table(seu_cbFilt$Phase) ``` ``` ## ## G1 G2M S ## 3713 2771 2361 ``` Cell Cycle Phase determined by *scran::cyclone()* ``` r table(seu_cbFilt$cyclon_Phase) ``` ``` ## ## G1 G2M S ## 8386 229 102 ``` --- ## Compare the two strategies Compare the two method: *Seurat* in row and *scran* in column ``` r table(seu_cbFilt$Phase, seu_cbFilt$cyclon_Phase) ``` ``` ## ## G1 G2M S ## G1 3447 95 45 ## G2M 2650 85 34 ## S 2289 49 23 ``` --- ## Compare the two strategies - *cyclone* assigned more cells to G1 phase. It is typically much more conservative. This is good for cells that have a low cycle rate i.e. Neurons. - *cyclone* takes much more time than *CellCycleScoring*. - It will likely be worth to process both methods and evaluate which results make more sense for your own dataset. --- class: inverse, center, middle # Detecting Doublets <html><div style='float:left'></div><hr color='#EB811B' size=1px width=720px></html> --- ## Detect doublets with Scrublet Doublets mean multiple cells clumped in the same single droplet. This is a problem: * A doublet may have many more UMIs and Genes in a single cell barcode than the overall population. * Multiple marker genes can be detected in doublets at the same time, even if they are mutually exclusive. Scrublet is a tool to detect doublets. * The GitHub [link](https://github.com/swolock/scrublet) * Original Paper on bioRxiv [link](https://www.biorxiv.org/content/10.1101/357368v1) * It's based on python. But we can use reticulate to invoke scrublet in R. --- ## Installing Scrublet We can install the scrublet easily using the Herper package. This uses reticulate to manage miniconda, a repository of tools from which we can install scrublet. ``` r library(reticulate) reticulate::py_install("scrublet") scr <- reticulate::import("scrublet") ``` --- ## Running scrublet First, we need the Seurat counts as an input for scrublet. ``` r mat <- GetAssayData(seu_cbFilt, assay = "RNA", slot = "counts") mat <- as.matrix(mat) ``` --- ## Running scrublet As scrublet is a python tool, we use slightly different nomenclature to what we are used to for R functions. ``` r # Loading the scrublet library scr <- import("scrublet") # Run scrublet scrub <- scr$Scrublet(t(mat)) # Extract scrublet results doublet <- scrub$scrub_doublets() names(doublet) <- c("doublet_score", "doublet") ``` --- ## Scrublet results Similar to the Cell Cycle scoring, we get a score and an assigment from scrublet. **Doublet score** ``` r summary(doublet$doublet_score) ``` ``` ## Min. 1st Qu. Median Mean 3rd Qu. Max. ## 0.00416 0.05194 0.08621 0.11890 0.14172 0.88750 ``` **Doublet Count** ``` r table(doublet$doublet) ``` ``` ## ## FALSE TRUE ## 8352 493 ``` --- ## Import Scrublet results into Seurat We can just assign our results into our Seurat object. ``` r seu_cbFilt[["doublet_score"]] <- doublet$doublet_score seu_cbFilt[["doublet"]] <- doublet$doublet ``` --- ## Assessing Doublets We can use violin plots to check our doublet predictions make sense. As expected out doublets (TRUE) have higher, doublet scores, UMIs (nCount), genes (nFeature) compared to singlets (FALSE). ``` r VlnPlot(seu_cbFilt, group.by = "doublet", features = c("doublet_score", "nCount_RNA", "nFeature_RNA"), pt.size = 0) ``` <!-- --> --- ## Assessing Doublets An alternative visualization of this is an X-Y plot comparing UMIs (nCount) and genes (nFeature). ``` r FeatureScatter(seu_cbFilt, feature1 = "nCount_RNA", feature2 = "nFeature_RNA", pt.size = 0.1, group.by = "doublet") ``` <!-- --> --- ## Other doublet removal methods - There are different software used for doublet removal in both R and Python (which you can use with reticulate). Follow this [link](https://www.sciencedirect.com/science/article/pii/S2405471220304592) for a good review about them. - DoubletFinder is a popular software in R. It all depends on your assumptions about your data. --- ## Other doublet removal methods - Scrublet: creates an artificial doublet from your data to compare with cells by summing the expression of 2 random cells. Uses raw data and can mistakenly identify cells with high depth as doublets. - DoubletFinder: creates an artificial doublet from your data by averaging the expression of 2 random cells. Uses normalized data and can reliably identify doublets regardless of RNA content. It can miss doublets with high RNA content. --- class: inverse, center, middle # Quality Assessment <html><div style='float:left'></div><hr color='#EB811B' size=1px width=720px></html> --- ## Quality Assessment There are many aspects to determining the quality of your scRNAseq dataset. You can see we have already made several plots to descirbe features of the data. These help guide additional QC steps. We will dig more into that later. For now we will show you several ways to plot the data and the various metrics we have to try and assess the state of our dataset. --- ## Distribution of variables in violin plot We can use violin plots to check the distribution of specific variables such as: + UMIs (*nCount_RNA*) per cell + Genes detected (*nFeature_RNA*) per cell + Ratio of UMIs of mitochondrial genes to nucleus genes (*percent.mt*) ``` r VlnPlot(seu_cbFilt, group.by = "dset", features = c("nCount_RNA", "nFeature_RNA", "percent.mt"), pt.size = 0) ``` --- ## Distribution of variables in violin plot If a bimodal distribution is identified, we need to check for any interference as it suggests that there could be multiple populations within our dataset. This could be biological (i.e very different cell types) or technical (doublets). We can see some evidence of bimodality in our dataset here. <!-- --> --- ## Evaluating interactions Scatter plots can be used to look for interactions between specific variables, like we did earlier. Here we compare UMIs and genes detected per cell: * UMIs (*nCount_RNA*) and the genes detected (*nFeature_RNA*) should be in high correlation * Doublets with high UMIs and high genes detected. <!-- --> --- ## Evaluating interactions We can use the same approach with different variables to compare between UMI counts and the percent of mitochondrial content: * Potential cell debris would show low UMI counts (*nCount_RNA*) and high percentage of mitochondrial genes (*percent.mt*) ``` r FeatureScatter(seu_cbFilt, feature1 = "nCount_RNA", feature2 = "percent.mt") ``` --- ## Evaluating interactions Comparison between UMI counts and the percent of mitochondrial content: * Potential cell debris would show low UMI counts (*nCount_RNA*) and high percentage of mitochondrial genes (*percent.mt*) <!-- --> --- ## Ridge plot Another plot you will also often see is a ridge/joy plot. These are evry similar to violins we have already made. * This plot would be applied to demonstrate a given variable and their corresponding score (e.g. doublet vs doublet_score). * It is applied to evaluate the difference between groups in the variable. * This plot is most widely applied in hash-tag determination of CITE-Seq. We could discuss more details in Section III. ``` r RidgePlot(seu_cbFilt, group.by = "doublet", features = c("doublet_score")) ``` --- ## Ridge plot Another plot you will also often see is a ridge/joy plot. These are evry similar to violins we have already made. * This plot would be applied to demonstrate a given variable and their corresponding score (e.g. doublet vs doublet_score). * It is applied to evaluate the difference between groups in the variable. * This plot is most widely applied in hash-tag determination of CITE-Seq. We could discuss more details in Section III. ``` ## Picking joint bandwidth of 0.0198 ``` <!-- --> --- class: inverse, center, middle # Filtering debris and doublets <html><div style='float:left'></div><hr color='#EB811B' size=1px width=720px></html> --- ## Filtering doublets and cell debris Once we have described and annotated our data, we can start to enact our quality control methods to filter out bad cells and correct confounding variables. Cell debris are with high percent.mt and low UMI counts. Generally, we set various cut-off on percent.mt: * In most scRNA cases, we set the *percent.mt > 10* (95% of overall population) * For several specific tissues with high oxygen consumption, like activated leukocytes or muscles, we set the *percent.mt > 25*. * For single-nuclei profiling, we should not get any UMIs originated from mitochondrial genes. We set the *percent.mt > 1*. The doublets detected by Scrublet shall also be removed at this step. --- ## Filtering doublets and cell debris How many cells will be removed: ``` r table(seu_cbFilt$doublet == "TRUE" | seu_cbFilt$percent.mt >= 10) ``` ``` ## ## FALSE TRUE ## 8088 757 ``` Filtering the cells: ``` r seu_filt <- subset(seu_cbFilt, subset = doublet == "FALSE" & percent.mt < 10) ``` --- ## Filtering doublets and cell debris - Distribution of variables before filtering <!-- --> --- ## Filtering doublets and cell debris - Distribution of variables after filtering 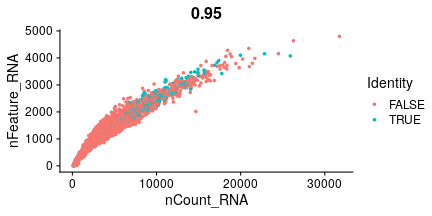<!-- --> --- ## Filtering doublets and cell debris - Other common quality filtering: - Gene content (e.g.: nFeature_RNA > 200 & nFeature_RNA < 2500) - UMI content (e.g. : nCount_RNA > 500 & nCount_RNA < 5000 ) - Removing all mitochondrial content - CITEseq: doublets and hashthag negative cells - Cells with a high ribosomal percentage --- class: inverse, center, middle # Regress out confounders <html><div style='float:left'></div><hr color='#EB811B' size=1px width=720px></html> --- ## Variance and confounders Though we have filtered out the most extreme problem cells we will likely still have confounders in our dataset. These are unexpected variables contribute to a high percent of the variances. - The *vars.to.regress* argument in *ScaleData()* allows us account for confounders. - Common potential confounders: percent.mt, doublet_score, cell cycle scores. ``` r # First let's replace NA values in the cyclon phase columns because ScaleData # doesn't like this cyclone_cols_numeric <- c("cyclon_G1Score", "cyclon_G2MScore", "cyclon_SScore") # For each numeric column, replace NA with 0 for (col_name in cyclone_cols_numeric) { seu_filt@meta.data[[col_name]][is.na(seu_filt@meta.data[[col_name]])] <- 0 } pot_conf <- c("percent.mt", "doublet_score", "cyclon_G1Score", "cyclon_SScore", "cyclon_G2MScore") seu_filt <- ScaleData(seu_filt, vars.to.regress = pot_conf) ``` --- ## Re-processing of data - It's important to re-run dimensionality reduction and re-cluster your cells once you've done filtering - Let's start by using data_proc() to quickly normalize and find variables in our data ``` r seu_filt <- data_proc(seu_filt) ``` --- ## Re-processing of data - Next, we will quickly re-run PCA and derive the elbow, saving the number of principal components to use in "pc" ``` r seu_filt <- RunPCA(seu_filt, assay = "RNA", npcs = 50) # Determine percent of variation associated with each PC pct <- seu_filt[["pca"]]@stdev/sum(seu_filt[["pca"]]@stdev) * 100 # Calculate cumulative percents for each PC cumu <- cumsum(pct) # Determine which PC exhibits cumulative percent greater than 90% and % # variation associated with the PC as less than 5 co1 <- which(cumu > 90 & pct < 5)[1] # Determine the difference between variation of PC and subsequent PC, last # point where change of % of variation is more than 0.1%. co2 <- sort(which((pct[1:length(pct) - 1] - pct[2:length(pct)]) > 0.1), decreasing = T)[1] + 1 pc <- min(co1, co2) ``` --- ## Re-processing of data - Now, we will perform non-linear dimensionality reduction with our previously calculated optimal pcs - After this we are ready to re-cluster ``` r seu_filt <- FindNeighbors(seu_filt, dims = 1:pc, reduction = "pca") seu_filt <- RunUMAP(seu_filt, dims = 1:pc, reduction = "pca") ``` --- ## A basic scRNAseq workflow  --- class: inverse, center, middle # Clustering <html><div style='float:left'></div><hr color='#EB811B' size=1px width=720px></html> --- ## Clustering - We will once more use the *FindClusters()* function in Seurat, but we will refine the resolution - This will give us an optimal number of clusters - To find out a better resolution, we test clustering with different resolutions and evaluate in a clustree. --- ## Identify clusters For comparison, we will generate clusters first with the default resolution of 0.5 ``` r seu_filt <- FindClusters(seu_filt, resolution = 0.5) ``` ``` ## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck ## ## Number of nodes: 8088 ## Number of edges: 267251 ## ## Running Louvain algorithm... ## Maximum modularity in 10 random starts: 0.8912 ## Number of communities: 13 ## Elapsed time: 0 seconds ``` ``` r seu_filt[["cluster_byDefault"]] <- seu_filt$seurat_clusters ``` --- ## Identify clusters Looking at the UMAP we can see good separation of clusters. But there may be room for improvement. ``` r DimPlot(seu_filt, group.by = "seurat_clusters", label = TRUE, pt.size = 0.2) + NoLegend() ``` 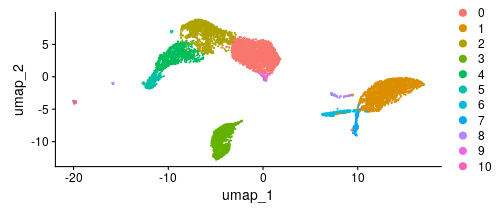<!-- --> --- ## Testing resolutions To identify the best resolution for each dataset we iteratively test potential resolutions using clustree. ``` r library(clustree) reso <- c(0.1, 0.2, 0.3, 0.4, 0.5, 0.6, 0.7, 0.8, 0.9, 1) reso_res <- lapply(1:length(reso), function(x, seu_filt, reso) { seu_filt <- FindClusters(seu_filt, resolution = reso[x]) clust <- setNames(seu_filt$seurat_clusters, Cells(seu_filt)) return(clust) }, seu_filt, reso) ``` ``` ## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck ## ## Number of nodes: 8088 ## Number of edges: 267251 ## ## Running Louvain algorithm... ## Maximum modularity in 10 random starts: 0.9578 ## Number of communities: 7 ## Elapsed time: 0 seconds ## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck ## ## Number of nodes: 8088 ## Number of edges: 267251 ## ## Running Louvain algorithm... ## Maximum modularity in 10 random starts: 0.9375 ## Number of communities: 9 ## Elapsed time: 0 seconds ## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck ## ## Number of nodes: 8088 ## Number of edges: 267251 ## ## Running Louvain algorithm... ## Maximum modularity in 10 random starts: 0.9202 ## Number of communities: 10 ## Elapsed time: 0 seconds ## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck ## ## Number of nodes: 8088 ## Number of edges: 267251 ## ## Running Louvain algorithm... ## Maximum modularity in 10 random starts: 0.9038 ## Number of communities: 12 ## Elapsed time: 0 seconds ## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck ## ## Number of nodes: 8088 ## Number of edges: 267251 ## ## Running Louvain algorithm... ## Maximum modularity in 10 random starts: 0.8912 ## Number of communities: 13 ## Elapsed time: 0 seconds ## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck ## ## Number of nodes: 8088 ## Number of edges: 267251 ## ## Running Louvain algorithm... ## Maximum modularity in 10 random starts: 0.8801 ## Number of communities: 13 ## Elapsed time: 0 seconds ## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck ## ## Number of nodes: 8088 ## Number of edges: 267251 ## ## Running Louvain algorithm... ## Maximum modularity in 10 random starts: 0.8679 ## Number of communities: 14 ## Elapsed time: 0 seconds ## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck ## ## Number of nodes: 8088 ## Number of edges: 267251 ## ## Running Louvain algorithm... ## Maximum modularity in 10 random starts: 0.8592 ## Number of communities: 17 ## Elapsed time: 0 seconds ## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck ## ## Number of nodes: 8088 ## Number of edges: 267251 ## ## Running Louvain algorithm... ## Maximum modularity in 10 random starts: 0.8503 ## Number of communities: 18 ## Elapsed time: 0 seconds ## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck ## ## Number of nodes: 8088 ## Number of edges: 267251 ## ## Running Louvain algorithm... ## Maximum modularity in 10 random starts: 0.8426 ## Number of communities: 18 ## Elapsed time: 0 seconds ``` ``` r names(reso_res) <- paste0("k", 1:length(reso)) ``` Some newer Macs have issues with *clustree*. Installing *tweenr* directly should help. ``` r remotes::install_github("thomasp85/tweenr") ``` --- ## Testing resolutions To identify the best resolution for each dataset we iteratively test potential resolutions ``` r k_tab <- do.call(cbind, reso_res) k_dat <- as.data.frame(k_tab) head(k_dat, 2) ``` ``` ## k1 k2 k3 k4 k5 k6 k7 k8 k9 k10 ## PBMC_8k_GCAGTTAGTTCCATGA-1 3 7 7 8 9 9 10 13 12 13 ## PBMC_8k_CCTTCCCTCATCTGTT-1 1 3 6 7 8 8 8 9 9 11 ``` --- ## Evaluate resolutions with clustree - The resolution from top to the bottom are *0.1,0.2,0.3,0.4,0.5,0.6,0.7,0.8,0.9,1* - To identify the better resolution + less cross talks between lineages + more clusters identified ``` r clustree(k_dat, prefix = "k", node_colour = "sc3_stability") ``` --- ## Evaluate resolutions with clustree - The resolution from top to the bottom are *0.1,0.2,0.3,0.4,0.5,0.6,0.7,0.8,0.9,1* * Let's try *resolution=0.3* first, which has the least cross-talk <!-- --> --- ## Clustering - optimized Lets retry clustering with our improved resolution. ``` r seu_filt <- FindClusters(seu_filt, resolution = 0.3) ``` ``` ## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck ## ## Number of nodes: 8088 ## Number of edges: 267251 ## ## Running Louvain algorithm... ## Maximum modularity in 10 random starts: 0.9202 ## Number of communities: 10 ## Elapsed time: 0 seconds ``` --- ## Clustering - optimized - We can see that we have slightly less clusters. We can now compare back to the original clustering, and there are some clusters that previously overlapped and are now classified as one. ``` r DimPlot(seu_filt, group.by = "seurat_clusters", label = TRUE, pt.size = 0.2) + NoLegend() + ggtitle("Optimized Clusters") ``` <!-- --> --- ## Clustering - default - We can see that we have slightly less clusters. We can now compare back to the original clustering, and there are some clusters that previously overlapped and are now classified as one. - Specifically cluster 2 and 6 are now cluster 1 ``` r DimPlot(seu_filt, group.by = "cluster_byDefault", label = TRUE, pt.size = 0.2) + NoLegend() + ggtitle("Default Clusters") ``` 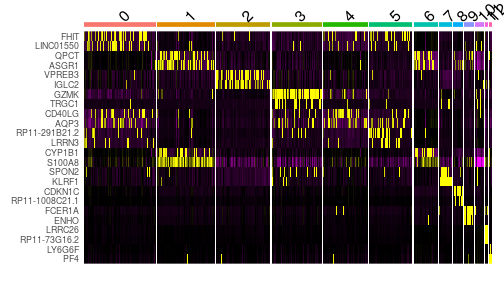<!-- --> --- ## Gene marker expression - We can plot the expression of LYZ onto our clusters using FeaturePlot() - We see that it is clearly present in cluster 1,6,7,9 - However, the expression of LYZ on new cluster 4 indicates that maybe cluster 4 is composed of 2 cell types ``` r FeaturePlot(seu_filt, features = "LYZ", pt.size = 0) ``` <!-- --> --- ## Clustering - optimized Lets retry clustering with 0.6 - which should yield more clusters than 0.5 ``` r seu_filt <- FindClusters(seu_filt, resolution = 0.6) ``` ``` ## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck ## ## Number of nodes: 8088 ## Number of edges: 267251 ## ## Running Louvain algorithm... ## Maximum modularity in 10 random starts: 0.8801 ## Number of communities: 13 ## Elapsed time: 0 seconds ``` --- ## Clustering - optimized - We can see that we have slightly more clusters than 0.3 and 0.5. We can now compare back to the original clustering, and we see cluster 10 is back, but now there's some extra clusters that don't visually make sense ``` r DimPlot(seu_filt, group.by = "seurat_clusters", label = TRUE, pt.size = 0.2) + NoLegend() + ggtitle("Optimized Clusters") ``` 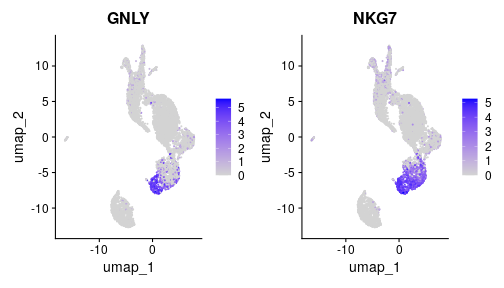<!-- --> --- ## Clustering - default - We can see that we have slightly more clusters than 0.3 and 0.5. We can now compare back to the original clustering, and we see cluster 10 is back, but now there's some extra clusters that don't visually make sense ``` r DimPlot(seu_filt, group.by = "cluster_byDefault", label = TRUE, pt.size = 0.2) + NoLegend() + ggtitle("Default Clusters") ``` <!-- --> --- ## Clustering - optimized - Let's try 0.4 to see if it rescues cluster 10 but unites original clusters 2 and 6 - In any case, if you suspect a cluster is made of different subtypes and can't obtain clear separation without forcing additional clusters, you can isolate (subset) the cluster later and re-cluster ``` r seu_filt <- FindClusters(seu_filt, resolution = 0.4) ``` ``` ## Modularity Optimizer version 1.3.0 by Ludo Waltman and Nees Jan van Eck ## ## Number of nodes: 8088 ## Number of edges: 267251 ## ## Running Louvain algorithm... ## Maximum modularity in 10 random starts: 0.9038 ## Number of communities: 12 ## Elapsed time: 0 seconds ``` --- ## Clustering - optimized - We now see that original clusters 2 and 6 are united in cluster 1, and original clusters 4 and 10 remain separated as clusters 4 and 9. ``` r DimPlot(seu_filt, group.by = "seurat_clusters", label = TRUE, pt.size = 0.2) + NoLegend() + ggtitle("Optimized Clusters") ``` <!-- --> --- ## Clustering - default - We now see that original clusters 2 and 6 are united in cluster 1, and original clusters 4 and 10 remain separated as clusters 4 and 9. ``` r DimPlot(seu_filt, group.by = "cluster_byDefault", label = TRUE, pt.size = 0.2) + NoLegend() + ggtitle("Default Clusters") ``` <!-- --> --- ## A basic scRNAseq workflow  --- ## Identify cluster-specific marker genes Once we have defined clusters, we want to know what distinguishes them. We can identify marker genes for each cluster by using *Seurat::FindAllMarker()*. In this case we are selecting only positive markers (only.pos=TRUE), genes that are found in 0.25 of cells and have a logFC 0.25 difference. ``` r markers <- FindAllMarkers(seu_filt, only.pos = TRUE, min.pct = 0.25, logfc.threshold = 0.25) ``` ``` r head(markers) ``` ``` ## p_val avg_log2FC pct.1 pct.2 p_val_adj cluster gene ## LEF1 0 2.3541627 0.658 0.165 0 0 LEF1 ## CCR7 0 2.2946188 0.659 0.168 0 0 CCR7 ## LDHB 0 1.3104935 0.939 0.607 0 0 LDHB ## RPL30 0 0.5992342 0.999 0.987 0 0 RPL30 ## RPL9 0 0.5829411 0.999 0.988 0 0 RPL9 ## RPL31 0 0.7571619 0.998 0.988 0 0 RPL31 ``` --- ## Identify cluster-specific marker genes We can review the results with a heatmap. We will first select the top 2 genes for each cluster by log2FC. ``` r top_genes <- markers %>% group_by(cluster) %>% slice_max(n = 2, order_by = avg_log2FC) head(top_genes) ``` ``` ## # A tibble: 6 x 7 ## # Groups: cluster [3] ## p_val avg_log2FC pct.1 pct.2 p_val_adj cluster gene ## <dbl> <dbl> <dbl> <dbl> <dbl> <fct> <chr> ## 1 3.52e-215 3.14 0.267 0.033 1.18e-210 0 RP11-291B21.2 ## 2 0 2.35 0.658 0.165 0 0 LEF1 ## 3 0 5.57 0.905 0.041 0 1 S100A12 ## 4 0 5.30 0.974 0.111 0 1 S100A8 ## 5 0 8.60 0.57 0.005 0 2 VPREB3 ## 6 0 8.59 0.45 0.015 0 2 IGLC2 ``` --- ## Identify cluster-specific marker genes - We can then use the *DoHeatmap()* from Seurat to plot just these genes - You can see here whether clusters have unique markers or if they overlap with other clusters ``` r DoHeatmap(seu_filt, features = top_genes$gene) + NoLegend() ``` ``` ## Warning in DoHeatmap(seu_filt, features = top_genes$gene): The following ## features were omitted as they were not found in the scale.data slot for the RNA ## assay: AAK1, CASP8 ``` <!-- --> --- class: inverse, center, middle # Evaluate known marker genes expression <html><div style='float:left'></div><hr color='#EB811B' size=1px width=720px></html> --- ## Known marker genes in PBMC data * T-cells + Naive CD4 T-cell: IL7R, and CCR7 + Memory CD4 T-cell: IL7R,S100A4 + CD8 T-cell: CD8A * B-cells: MS4A1 * Monocytes + CD14+ Monocyte: CD14,LYZ + FCGR3A+ Monocyte: FCGR3A,MS4A7 * Dendritic cells (DCs): FCER1A,CST3 * Nature Killer cells (NKs): GNLY,NKG7 * Platelets: PPBP --- ## Evaluate marker gene expressions We can use the *FeaturePlot()* function to look at the expression of speicfic genes i.e. Naive CD4 T-cell ``` r known_marker <- c("IL7R", "CCR7") FeaturePlot(seu_filt, features = known_marker) ``` <!-- --> --- ## Evaluate marker gene expressions Here is another example with NK cells. ``` r known_marker <- c("GNLY", "NKG7") FeaturePlot(seu_filt, features = known_marker) ``` <!-- --> --- ## Match markers to clusters We can also check the expression of markers in each cluster. First, we need to get our count data and our full list of markers. ``` r known_marker <- c("IL7R", "CCR7", "S100A4", "CD8A", "MS4A1", "CD14", "LYZ", "FCGR3A", "MS4A7", "FCER1A", "CST3", "GNLY", "NKG7", "PPBP") mat <- GetAssayData(seu_filt, assay = "RNA", slot = "data") mat <- mat[known_marker, ] mat <- as.matrix(mat) ``` --- ## Match markers to clusters Next we need to extract out the counts per cluster for our genes of interest. Here we use an lapply to iterate over every cluster, extracting counts and getting the average for each gene of interest. ``` r clust <- unique(seu_filt$seurat_clusters) clust <- as.character(clust) avgExp_byClust <- lapply(clust, function(clust, seu, known) { sub <- subset(seu, subset = seurat_clusters == clust) mat <- GetAssayData(sub, assay = "RNA", slot = "data") mat <- mat[known, ] mat <- as.matrix(mat) avg <- rowMeans(mat) return(avg) }, seu_filt, known_marker) names(avgExp_byClust) <- paste0("C", clust) ``` --- ## Match markers to clusters Finally we get to draw a heatmap with *pheatmap()*. We need a matrix for this, so we first stick our results from each cluster together. ``` r library(pheatmap) avgExp_mat <- do.call(cbind, avgExp_byClust) pheatmap(avgExp_mat, scale = "row") ``` <!-- --> --- ## Assign cell types by clusters We can then manually annotate the clusters based on these markers. We do this manually based on the average expression of markers in the cluster. We will show you how to automate this later. ``` r seu_filt[["cellType_byClust"]] <- NA seu_filt$cellType_byClust[seu_filt$seurat_clusters %in% c(0, 5)] <- "Naive CD4+ T cells" seu_filt$cellType_byClust[seu_filt$seurat_clusters %in% c(3)] <- "Memory CD4+ T cells" seu_filt$cellType_byClust[seu_filt$seurat_clusters %in% c(4)] <- "CD8+ T cells" seu_filt$cellType_byClust[seu_filt$seurat_clusters %in% c(6)] <- "NK cells" seu_filt$cellType_byClust[seu_filt$seurat_clusters %in% c(7, 10)] <- "DC" seu_filt$cellType_byClust[seu_filt$seurat_clusters %in% c(8)] <- "FCGR3A+ Monocytes" seu_filt$cellType_byClust[seu_filt$seurat_clusters %in% c(2)] <- "B cells" seu_filt$cellType_byClust[seu_filt$seurat_clusters %in% c(1)] <- "CD14+ Monocytes" seu_filt$cellType_byClust[seu_filt$seurat_clusters %in% c(11)] <- "Platelets" seu_filt$cellType_byClust[is.na(seu_filt$cellType_byClust)] <- "Unspecified" ``` --- ## Assign cell types by clusters Cell numbers in each cell type ``` r table(seu_filt$cellType_byClust) ``` ``` ## ## B cells CD14+ Monocytes CD8+ T cells DC ## 1142 1627 966 268 ## FCGR3A+ Monocytes Memory CD4+ T cells NK cells Naive CD4+ T cells ## 208 1002 321 2346 ## Platelets Unspecified ## 54 154 ``` --- ## Assign cell types by clusters We can also display seurat clusters and cell types in UMAP. ``` r DimPlot(seu_filt, group.by = c("seurat_clusters", "cellType_byClust"), label = F, pt.size = 0.2) ``` <!-- --> --- class: inverse, center, middle # Loupe Browser <html><div style='float:left'></div><hr color='#EB811B' size=1px width=720px></html> --- ## Loupe Browser for visualization .pull-left[ The Loupe browser is a tool for visualization of Cell Ranger cloupe files. It provides t-sne visualization of your single-cell data alongside sample/cell information as well as methods to test and visualize changes in gene expression. Loupe browser can be freely downloaded from the 10x website. ] .pull-right[ <div align="center"> <img src="imgs/loupeDownload.png" alt="igv" height="400" width="350"> </div> ] --- ## Creating Loupe files We can export our seurat objects to a cloupe file using the loupeR package from 10X. This package is available on their github. Once installed we need agree to the license by running the setup function. ``` r remotes::install_github("10xGenomics/loupeR") loupeR::setup() ``` --- ## Creating Loupe files We can then create a loupe file from our seurat object really easily using the *create_loupe_from_seurat()* function. The exported Loupe file will contain all dsicrete annotations we have added to our Seurat object i.e. Cell Cycle Phase, Cluster etc. ``` r library(loupeR) create_loupe_from_seurat(seu_filt, output_dir = "loupe", output_name = "seu_filt") ``` --- ## Loupe Browser for visualization .pull-left[ Having downloaded the Loupe browser we can load our cloupe files directly in and rapidly visually interrogate our data. We have a full guide into how to use and work with Loupe Browser [here](https://rockefelleruniversity.github.io/LoupeBrowser/) ] .pull-right[ <div align="center"> <img src="imgs/loupeFront.png" alt="igv" height="300" width="400"> </div> ] --- ## Further reading We have based our workflow on the [Seurat vignettes](https://satijalab.org/seurat/articles/get_started.html) and [OSCA book](https://bioconductor.org/books/release/OSCA/) from Bioconductor. UMAPS: * [Data Science blog post](https://towardsdatascience.com/dimensionality-reduction-for-data-visualization-pca-vs-tsne-vs-umap-be4aa7b1cb29) * [Bioinformatic presentation - Babraham Institute](https://www.bioinformatics.babraham.ac.uk/training/10XRNASeq/Dimension%20Reduction.pdf) * [Limitations](https://journals.plos.org/ploscompbiol/article?id=10.1371/journal.pcbi.1011288) - [Loupe Broswer Download](https://www.10xgenomics.com/products/loupe-browser/downloads)